9 0 5 7 8
论文已发表
注册即可获取德孚的最新动态
IF 收录期刊
- 2.6 Breast Cancer (Dove Med Press)
- 3.9 Clin Epidemiol
- 3.3 Cancer Manag Res
- 3.9 Infect Drug Resist
- 3.6 Clin Interv Aging
- 4.8 Drug Des Dev Ther
- 2.8 Int J Chronic Obstr
- 8.0 Int J Nanomed
- 2.3 Int J Women's Health
- 3.2 Neuropsych Dis Treat
- 4.0 OncoTargets Ther
- 2.2 Patient Prefer Adher
- 2.8 Ther Clin Risk Manag
- 2.7 J Pain Res
- 3.3 Diabet Metab Synd Ob
- 4.3 Psychol Res Behav Ma
- 3.4 Nat Sci Sleep
- 1.9 Pharmgenomics Pers Med
- 3.5 Risk Manag Healthc Policy
- 4.5 J Inflamm Res
- 2.3 Int J Gen Med
- 4.1 J Hepatocell Carcinoma
- 3.2 J Asthma Allergy
- 2.3 Clin Cosmet Investig Dermatol
- 3.3 J Multidiscip Healthc

已发表论文
对检测 A2059G 突变以鉴定阿奇霉素高耐淋球菌的分子学方法的准确性评估:系统综述和 Meta 分析
Authors Wang F, Liu JW, Liu HY, Huang J, Chen SC, Chen XS, Yin YP
Received 13 August 2018
Accepted for publication 14 November 2018
Published 28 December 2018 Volume 2019:12 Pages 95—104
DOI https://doi.org/10.2147/IDR.S183754
Checked for plagiarism Yes
Review by Single-blind
Peer reviewers approved by Dr Colin Mak
Peer reviewer comments 2
Editor who approved publication: Dr Joachim Wink
目的:淋病奈瑟菌(Neisseria
gonorrhoeae ,淋球菌)对阿奇霉素的耐药已成为全球主要的公共卫生问题,并且高水平的耐药株也经常出现。淋球菌对阿奇霉素的高水平耐药常被认为与其 23S rRNA 的 2059(大肠杆菌编号)位置上的等位基因突变有关。如今对高耐株的鉴定仍然主要依赖于传统的琼脂稀释方法和 E-test 法。本研究旨在评估用检测 A2059G 突变来鉴定对阿奇霉素高耐淋球菌的分子学方法的准确性,进而确定 A2059G 突变和阿奇霉素高耐之间的关系。
方法:两名研究人员独立检索六个数据库的文献,搜集上述分子学检测方法检测到的阿奇霉素高水平耐药株的研究结果。固定效应模型用于估计合并的敏感性、特异性、阳性预测值和阴性预测值及其相应的 95% 置信区间(CIs)。生成总结受试者工作特征(sROC)曲线,并通过曲线下面积(AUC)来估计分子学方法的总体表现。Deeks 测试用以评估潜在的发表偏倚。
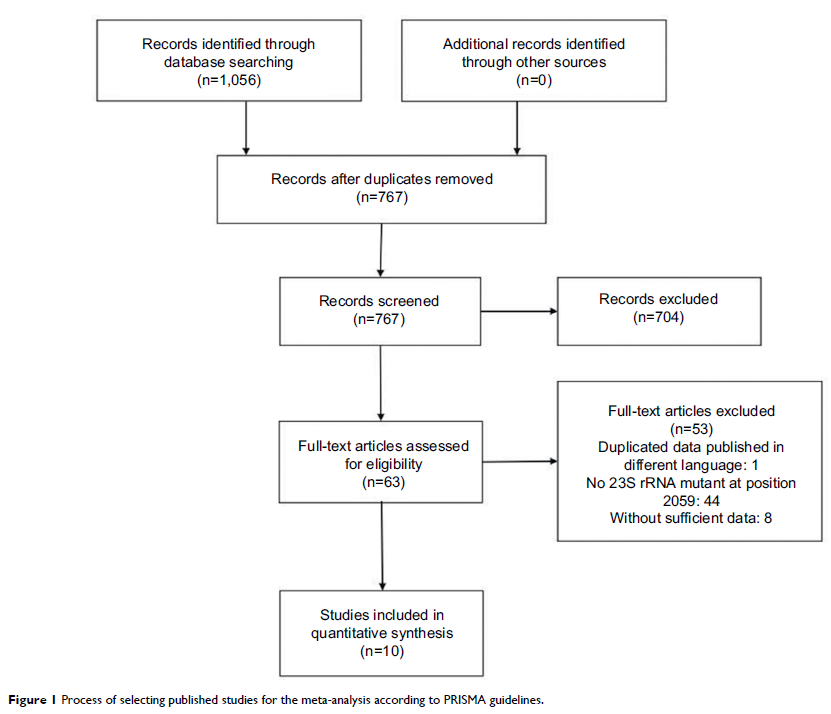
结果:10 项相关研究纳入 Meta 分析以评估分子检测方法的总体准确性。分子生物学检测方法的合并敏感性为 97.8%(95% CI:93.8%〜99.5%),合并特异性为 99.1%(95% CI:98.0%〜99.7%),合并阴性预测值为 96.4%(95% CI:91.9%〜98.8%),合并阴性预测值为 99.5%(95% CI:98.5%〜99.9%)。通过 sROC 曲线得出的 AUC 为 0.99,这表明通过检测 A2059G 突变的分子生物学检测方法对鉴定阿奇霉素高耐株具有相对高的整体准确性。发表偏倚存在统计学差异。
结论:淋球菌 23S rRNA 的 A2059G 突变是造成对阿奇霉素高水平耐药的关键因素。纳入本研究的分子学检测方法较高的敏感性和特异性,可能造成了数据分析模型的发表偏倚。因此,推荐该方法在商品化后用于临床,这必将帮助临床医生为淋病感染患者给予精准治疗。
关键词:淋病奈瑟菌;阿奇霉素;抗生素耐药;系统综述
方法:两名研究人员独立检索六个数据库的文献,搜集上述分子学检测方法检测到的阿奇霉素高水平耐药株的研究结果。固定效应模型用于估计合并的敏感性、特异性、阳性预测值和阴性预测值及其相应的 95% 置信区间(CIs)。生成总结受试者工作特征(sROC)曲线,并通过曲线下面积(AUC)来估计分子学方法的总体表现。Deeks 测试用以评估潜在的发表偏倚。
结果:10 项相关研究纳入 Meta 分析以评估分子检测方法的总体准确性。分子生物学检测方法的合并敏感性为 97.8%(95% CI:93.8%〜99.5%),合并特异性为 99.1%(95% CI:98.0%〜99.7%),合并阴性预测值为 96.4%(95% CI:91.9%〜98.8%),合并阴性预测值为 99.5%(95% CI:98.5%〜99.9%)。通过 sROC 曲线得出的 AUC 为 0.99,这表明通过检测 A2059G 突变的分子生物学检测方法对鉴定阿奇霉素高耐株具有相对高的整体准确性。发表偏倚存在统计学差异。
结论:淋球菌 23S rRNA 的 A2059G 突变是造成对阿奇霉素高水平耐药的关键因素。纳入本研究的分子学检测方法较高的敏感性和特异性,可能造成了数据分析模型的发表偏倚。因此,推荐该方法在商品化后用于临床,这必将帮助临床医生为淋病感染患者给予精准治疗。
关键词:淋病奈瑟菌;阿奇霉素;抗生素耐药;系统综述