108552
论文已发表
注册即可获取德孚的最新动态
IF 收录期刊
- 3.4 Breast Cancer (Dove Med Press)
- 3.2 Clin Epidemiol
- 2.6 Cancer Manag Res
- 2.9 Infect Drug Resist
- 3.7 Clin Interv Aging
- 5.1 Drug Des Dev Ther
- 3.1 Int J Chronic Obstr
- 6.6 Int J Nanomed
- 2.6 Int J Women's Health
- 2.9 Neuropsych Dis Treat
- 2.8 OncoTargets Ther
- 2.0 Patient Prefer Adher
- 2.2 Ther Clin Risk Manag
- 2.5 J Pain Res
- 3.0 Diabet Metab Synd Ob
- 3.2 Psychol Res Behav Ma
- 3.4 Nat Sci Sleep
- 1.8 Pharmgenomics Pers Med
- 2.0 Risk Manag Healthc Policy
- 4.1 J Inflamm Res
- 2.0 Int J Gen Med
- 3.4 J Hepatocell Carcinoma
- 3.0 J Asthma Allergy
- 2.2 Clin Cosmet Investig Dermatol
- 2.4 J Multidiscip Healthc

基于生物信息学分析构建 COPD 血浆中潜在的 miRNA-mRNA 调控网络
Authors Zhu M, Ye M, Wang J, Ye L, Jin M
Received 31 March 2020
Accepted for publication 18 August 2020
Published 10 September 2020 Volume 2020:15 Pages 2135—2145
DOI https://doi.org/10.2147/COPD.S255262
Checked for plagiarism Yes
Review by Single anonymous peer review
Peer reviewer comments 2
Editor who approved publication: Dr Richard Russell
背景:慢性阻塞性肺疾病(COPD)已经成为全球发病率和死亡率较高的疾病之一。越来越多的研究表明,异常表达的微小 RNA(miRNA)参与了 COPD 的发生发展过程。然而,尚缺乏对 COPD 血浆中 miRNA-mRNA 综合调控网络的构建。
方法:从公共基因芯片数据库(GEO)下载微阵列数据集 GSE24709,GSE61741 和 GSE31568,并使用 GEO2R 在线分析工具筛选出 COPD 患者和正常人血浆之间的差异表达 miRNA(DEM),选择在上述三个数据集中具有一致变化的 miRNA 作为候选 DEM。候选 DEM 的潜在上游转录因子和下游靶基因分别由 FunRich 软件和 miRNet 数据库预测。然后,使用 DAVID 数据库对下游靶基因进行基因本体论(GO)分析和京都基因与基因组百科全书(KEGG)分析,使用 STRING 数据库和 Cytoscape 软件构建蛋白质-蛋白质相互作用(PPI)网络和 DEM-核心基因网络。最后,GSE56768 用于验证核心基因的表达。
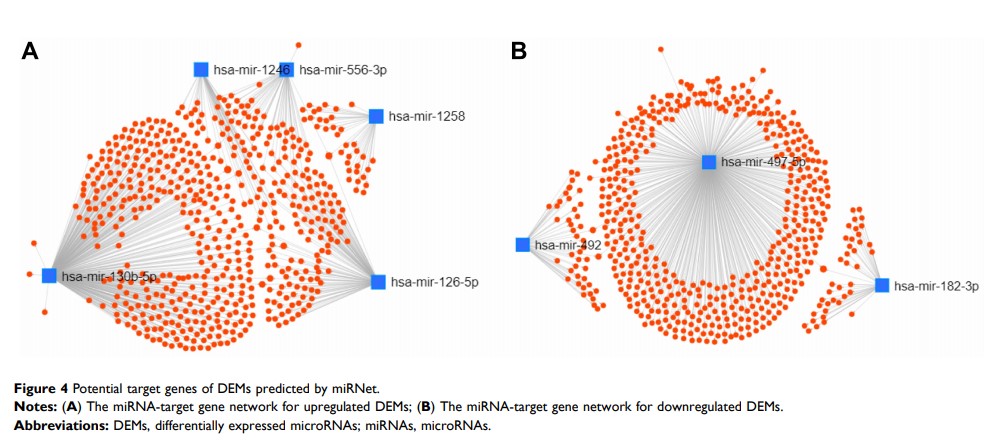
结果:对 GSE24709,GSE61741 和 GSE31568 三个数据集进行 DEM 分析后共挑选出 9 个(6 个上调和 3 个下调)共同 DEMs。FunRich 软件预测 SP1 可以调控大多数下调的 DEMs,而 YY1 和 E2F1 可以同时调控上调和下调的 DEMs。通过 miRNet 数据库预测了 DEMs 有 1139 个靶基因,其中上调和下调的 DEMs 分别有 596 个和 543 个靶基因。这些靶基因主要富集在 PI3K/Akt,mTOR 和自噬等信号通路。通过 DEM-核心基因网络构建,发现大多数核心基因都可以被 miR-497-5p,miR-130b-5p 和 miR-126-5p 调节。在前 12 个核心基因中,MYC 和 FOXO1 的表达与在 GSE56768 数据集中的表达一致。
结论:在这项研究中,首次在 COPD 血浆中构建了潜在的 miRNA–mRNA 调控网络,这可能为 COPD 的发病机制和治疗提供新的见解。
关键词:微小RNA(miRNA);慢性阻塞性肺疾病(COPD);生物信息学分析; miRNA–mRNA 调控网络