110932
论文已发表
注册即可获取德孚的最新动态
IF 收录期刊
- 3.4 Breast Cancer (Dove Med Press)
- 3.2 Clin Epidemiol
- 2.6 Cancer Manag Res
- 2.9 Infect Drug Resist
- 3.7 Clin Interv Aging
- 5.1 Drug Des Dev Ther
- 3.1 Int J Chronic Obstr
- 6.6 Int J Nanomed
- 2.6 Int J Women's Health
- 2.9 Neuropsych Dis Treat
- 2.8 OncoTargets Ther
- 2.0 Patient Prefer Adher
- 2.2 Ther Clin Risk Manag
- 2.5 J Pain Res
- 3.0 Diabet Metab Synd Ob
- 3.2 Psychol Res Behav Ma
- 3.4 Nat Sci Sleep
- 1.8 Pharmgenomics Pers Med
- 2.0 Risk Manag Healthc Policy
- 4.1 J Inflamm Res
- 2.0 Int J Gen Med
- 3.4 J Hepatocell Carcinoma
- 3.0 J Asthma Allergy
- 2.2 Clin Cosmet Investig Dermatol
- 2.4 J Multidiscip Healthc

已发表论文
基于大样本数据分析的一种新型肝细胞癌生存风险评估模型构建和应用
Authors Zheng YJ, Liu YL, Zhao SF, Zheng ZT, Shen CY, An L, Yuan YL
Received 25 July 2018
Accepted for publication 25 October 2018
Published 21 November 2018 Volume 2018:10 Pages 6079—6096
DOI https://doi.org/10.2147/CMAR.S181396
Checked for plagiarism Yes
Review by Single-blind
Peer reviewers approved by Dr Justinn Cochran
Peer reviewer comments 2
Editor who approved publication: Dr Ahmet Emre Eskazan
背景:肝细胞癌(HCC)是一种高死亡率的原发性肝癌,发病率在世界范围内逐年增加,而目前缺少有效的诊断方法和预后生物标志物。
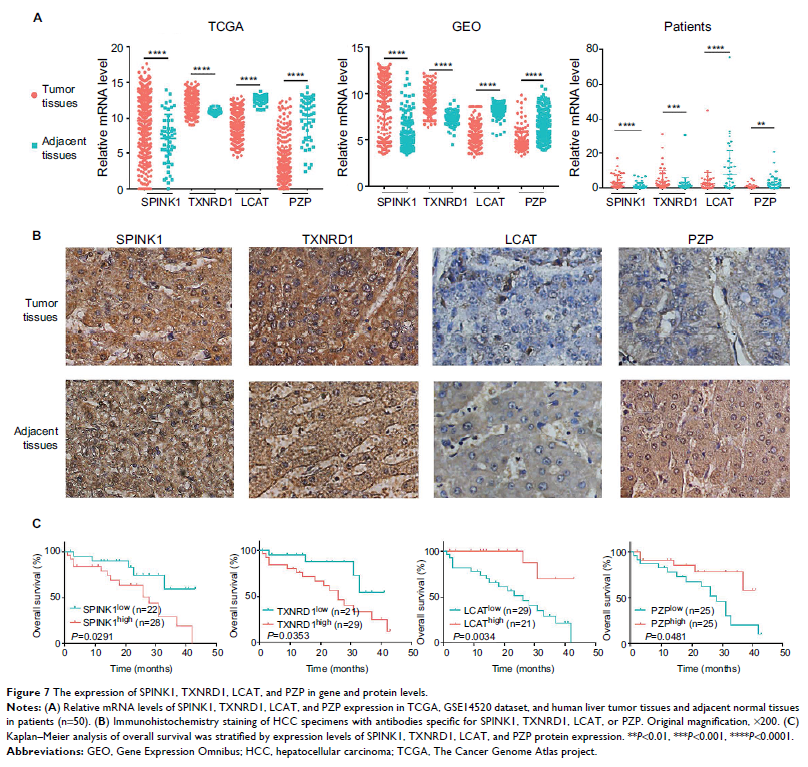
材料和方法:从 GEO 数据库收集 149 对 HCC 组织的基因表达谱及相关临床数据,进而筛选 HCC 和配对癌旁正常组织之间的差异表达基因(DEGs),并对 DEGs 使用 GO、KEGG 进行通路富集和 PPI 网络分析。接下来,利用 TCGA 数据库对 DEGs 进行单因素分析和 Cox 比例风险回归分析并构建 HCC 生存风险评估模型,并利用 GSE14520 数据库验证该模型。我们进一步通过 TCGA,GEO 和 qPCR 对风险模型中的关键分子 mRNA 表达进行了检测,并使用免疫组化证实了它们蛋白水平的表达。同时,我们利用 TCGA 数据库初步探究了这些基因表达与 DNA 甲基化状态的关系。
结果:从三个 GEO 数据集中筛选得到了 98 个 DEGs,其中有 13 个上调基因和 85 个下调基因; GO 和 KEGG 注释通路分析表明 DEGs 具有重要生物学特征; PPI 网络分析表明 DEGs 之间关系密切,该网络包括了 64 个节点和 115 个边缘。我们利用 TCGA 数据库对 DEGs 分析并成功建立了基于 4 个基因(SPINK1 ,TXNRD1 ,LCAT 和 PZP )表达的 HCC 生存风险评估模型,同时在 GSE14520 中得到了很好的验证。TCGA,GEO 和临床样本检测均证实这 4 个基因在 HCC 中具有一致的差异表达。TCGA 数据分析进一步提示,这 4 个基因的表达可能与 DNA 甲基化调控密切相关。
结论:本研究首次基于配对大样本数据分析构建了一种新型肝细胞癌生存风险评估模型,用于预测 HCC 的预后。风险模型中的 4 个生物标志物分析不仅能提示 HCC 发展的分子机制,更为肝癌的治疗策略提供新的思路。
关键词:肝细胞癌,GEO,TCGA,生物标志物,差异表达基因
材料和方法:从 GEO 数据库收集 149 对 HCC 组织的基因表达谱及相关临床数据,进而筛选 HCC 和配对癌旁正常组织之间的差异表达基因(DEGs),并对 DEGs 使用 GO、KEGG 进行通路富集和 PPI 网络分析。接下来,利用 TCGA 数据库对 DEGs 进行单因素分析和 Cox 比例风险回归分析并构建 HCC 生存风险评估模型,并利用 GSE14520 数据库验证该模型。我们进一步通过 TCGA,GEO 和 qPCR 对风险模型中的关键分子 mRNA 表达进行了检测,并使用免疫组化证实了它们蛋白水平的表达。同时,我们利用 TCGA 数据库初步探究了这些基因表达与 DNA 甲基化状态的关系。
结果:从三个 GEO 数据集中筛选得到了 98 个 DEGs,其中有 13 个上调基因和 85 个下调基因; GO 和 KEGG 注释通路分析表明 DEGs 具有重要生物学特征; PPI 网络分析表明 DEGs 之间关系密切,该网络包括了 64 个节点和 115 个边缘。我们利用 TCGA 数据库对 DEGs 分析并成功建立了基于 4 个基因(SPINK1 ,TXNRD1 ,LCAT 和 PZP )表达的 HCC 生存风险评估模型,同时在 GSE14520 中得到了很好的验证。TCGA,GEO 和临床样本检测均证实这 4 个基因在 HCC 中具有一致的差异表达。TCGA 数据分析进一步提示,这 4 个基因的表达可能与 DNA 甲基化调控密切相关。
结论:本研究首次基于配对大样本数据分析构建了一种新型肝细胞癌生存风险评估模型,用于预测 HCC 的预后。风险模型中的 4 个生物标志物分析不仅能提示 HCC 发展的分子机制,更为肝癌的治疗策略提供新的思路。
关键词:肝细胞癌,GEO,TCGA,生物标志物,差异表达基因